Studie Biologie: Geschmacksrezeptoren

Idee
Nachweisen des Bittergeschmacksrezeptor für PTC
Im Rahmen eines Workshops an der FH Wels wurde ein Verfahren zum Nachweis der Fähigkeit, einen bestimmten Stoff wahrzunehmen, angewendet. PTC ist ein Bitterstoff, der nur von manchen Leuten geschmeckt werden kann, da dafür ein gewisser Bittergeschmacksrezeptor notwendig ist. Etwa 70% der europäischen Bevölkerung haben das dazugehörige Gen. Es wird dominant vererbt.
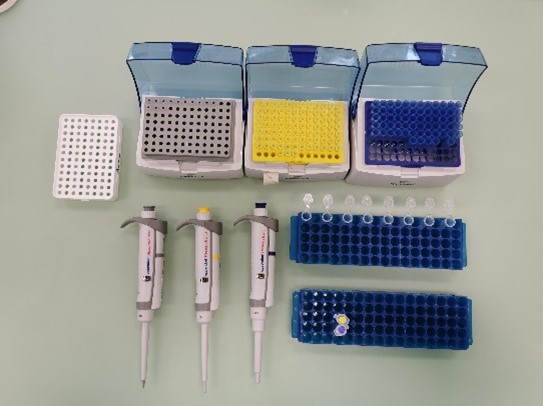
Es wurde mit drei unterschiedlichen Pipetten gearbeitet, welche je ein anderes Volumen hatten, um Flüssigkeiten auf µl genau aufzunehmen und in die PP-Röhrchen zu pipettieren.
Schritte
- DNA-Isolierung
- Polymerase-Kettenreaktion (PCR):
- Restriktionsverdau
- Gelelektrophorese
- Auswertung und Interpretation
DNA-Isolierung
Als Erstes wird der Mund für 30 Sekunden mit einer NaCl-Lösung (= Salzlösung), welche sich in einem PP-Röhrchen befindet, durchgespült. Daraufhin wird die Salzlösung zurück in das PP-Röhrchen gespuckt und vorsichtig geschwenkt, um die Zellen aufzuschwemmen, welche sich am Boden festgesetzt haben. Danach wird eine Pipette benutzt um 1000 µl der Lösung in ein 1,5 ml Eppi zu überführen.


Polymerase-Kettenreaktion (PCR):
Funktionsweise einer PCR
Um die DNA zu verdoppeln, wird die Lösung mit DNA-Doppelsträngen im ersten Schritt auf 100 °C erhitzt, um die Wasserstoffbrückenbindungen zu lösen und DNA-Einzelstränge zu erhalten. Daraufhin wird die Lösung je nach Primer in kurzer Zeit auf 50-70 °C erhitzt, damit sich die Primer an die DNA-Einzelstränge hängen. Die Primer dienen dazu, dass die Polymerase 3´-Enden haben, wo sie anfangen können zu arbeiten. Damit die Polymerase besonders gut arbeiten kann, wird die Lösung auf 72 °C erhitzt. Diese Polymerase platziert dann die jeweils passenden Nukleotide, welche in der Lösung enthalten sind, um wieder einen DNA-Doppelstrang zu erhalten. Dieser gesamte Vorgang wird Zyklus genannt und kann so oft wiederholt werden, bis die gewünschte Menge an DNA enthalten ist. Wie oft solch ein Zyklus durchgeführt wurde, gibt der CT-Wert an.
Vorbereitung für PCR
Zur Vorbereitung der PCR pipettiert man in einer 0,2ml PCR:
- 8 µl destilliertes H2O
- 2 µl Nukleosidtriphosphate (NTPs)
- 2 µl Primer PTC1
- 2 µl Primer PTC2
- 1 µl Polymerase
- 1 µl der eigenen DNA
- 4 µl PCR-Puffer

Restriktionsverdau

Gelelektrophorese

1) Nach der Beendigung des Restriktionsverdaus wurde die Probe aus dem Heizblock entnommen und 3µl Auftragspuffer dazugegeben. Gleich darauf wurden in die Probentasche (Abb. 8) auf der linken Seite 5µl des 50bp (=Basenpaare) Längenmarkers geladen.

Auswertung
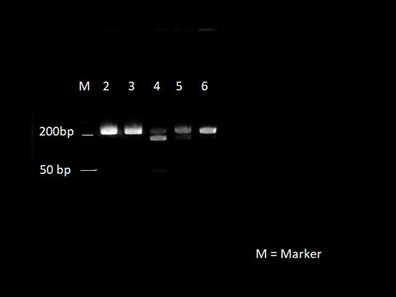
Die Testpersonen 2 & 3 & 6 sind Non-Taster (homozygot rezessiv), welches bedeutet, dass sie den Bitterstoff PTC nicht schmecken. Hingegen sind die Testpersonen 4 & 5 heterozygote Taster. Bei den Personen 2, 3 und 6 wird nicht geschnitten aufgrund der Mutation und deshalb ist jeweils nur eine Bande zu sehen. Aber bei den Personen 4 und 5 sind 3 Banden zu erkennen, da hier einmal geschnitten und einmal nicht geschnitten wird.
Interpretation
Im Gegensatz zum Europäischen Durchschnitts, welcher, wie im ersten Punkt erwähnt wurde, bei 70% Taster liegt, ergibt unser Ergebnis eine Mehrheit von Non-Tastern. Dies könnte daran liegen, dass die Stichprobengröße zu gering war. Ein weiterer Grund könnte sein, dass der Restriktionsverdau nicht funktioniert hat. Dem entgegenzuhalten ist das Ergebnis der anderen Gruppen, welche dem Durchschnitt entsprachen.